Menantang dominasi Google DeepMind dalam biologi komputasi, para peneliti di Harvard Medical School telah meluncurkan popEVE, model kecerdasan buatan baru yang dirancang untuk mendiagnosis penyakit genetik langka dengan spesifisitas yang ditingkatkan.
Diterbitkan hari ini di Nature Genetics, alat ini mengintegrasikan data populasi manusia untuk secara drastis mengurangi prediksi positif palsu, sebuah kelemahan yang terus-menerus terjadi pada model yang ada seperti AlphaMissense.
Dengan mengkalibrasi tingkat keparahan varian di seluruh proteom, popEVE berhasil mengidentifikasi 123 kandidat gen baru. untuk gangguan perkembangan, menawarkan terobosan diagnostik bagi pasien yang masih belum terselesaikan meskipun telah dilakukan pengujian ekstensif.
popEVE Bertujuan untuk Memecahkan Masalah Positif Palsu
Meskipun pengurutan genom berkembang pesat dalam lingkungan klinis, hasil diagnostik untuk kelainan genetik langka masih sangat rendah, dengan beberapa kelompok yang melihat hanya 25% dari sampel yang menerima diagnosis genetik definitif.
Dokter sering dihadapkan dengan beragam “Variant of Uncertain Significance”(VUS), perubahan genetik yang dampaknya terhadap kesehatan manusia tidak diketahui.
Keambiguan ini menciptakan hambatan diagnostik, sehingga mengidentifikasi varian spesifik yang bertanggung jawab atas kondisi pasien menjadi upaya yang memakan waktu dan sering kali sia-sia. Penafsiran saat ini sering kali gagal membedakan antara varian yang menyebabkan gangguan parah pada masa kanak-kanak dan varian dengan efek ringan yang hanya muncul di kemudian hari, yang merupakan perbedaan penting dalam perawatan anak.
Menurut makalah penelitian, popEVE mengatasi kesenjangan presisi ini dengan menerapkan ambang batas yang lebih ketat untuk patogenisitas. Dalam pengujiannya, model ini menunjukkan penurunan drastis dalam prediksi positif palsu pada populasi umum, dengan menandai hanya 11% individu sebagai pembawa varian virus yang parah.
Tingkat spesifisitas ini merupakan peningkatan yang nyata dibandingkan alat canggih yang sudah ada; misalnya, AlphaMissense dari Google DeepMind mengkategorikan sekitar 44% populasi umum membawa varian yang sama parahnya pada ambang batas ingatan yang sebanding. Dengan menyaring kebisingan ini, popEVE memungkinkan dokter untuk fokus pada varian yang paling mungkin menjadi penyebab.
Kemanjuran model ini divalidasi secara ketat pada metakohort yang terdiri dari 31.058 pasien dengan gangguan perkembangan parah (SDD), yang bersumber dari studi Deciphering Developmental Disorders (DDD), GeneDx, dan Radboud University Medical Center.
Dalam kumpulan data yang luas ini, Ambang batas tingkat keparahan tingkat kepercayaan tinggi popEVE (ditetapkan pada-5.056) mengungkapkan pengayaan varian patogen sebesar 15 kali lipat – lima kali lebih tinggi dibandingkan metode terkemuka lainnya seperti PrimateAI-3D. Kekuatan statistik ini memungkinkan model tersebut berhasil memberikan diagnosis untuk sekitar sepertiga kasus yang sebelumnya tidak dapat dijelaskan berdasarkan protokol pengujian standar.
Mungkin yang paling signifikan dalam bidang genetika medis adalah kemampuan model untuk mengungkap hubungan penyakit yang benar-benar baru. Analisis ini mengidentifikasi 123 kandidat gen baru yang terkait dengan gangguan perkembangan, 119 di antaranya dapat diidentifikasi pada tingkat varian tunggal.
Model seluruh protein untuk genetika penyakit manusia
(Sumber: Alam – CC BY-NC-ND 4.0)
Yang perlu diperhatikan, 31 gen ini ditemukan menggunakan varian missense saja – sebuah kategori mutasi yang biasanya memerlukan data Loss-of-Function (LoF) yang menguatkan agar dapat dianggap diagnostik. Kemampuan ini menunjukkan bahwa popEVE dapat mendeteksi sinyal patogen yang terlewatkan oleh metode berbasis pengayaan tradisional.
Validasi terhadap temuan ini sudah membuahkan hasil klinis. Sejak awal penelitian ini, 25 dari 123 kandidat gen baru telah dikonfirmasi secara independen oleh laboratorium lain dan secara resmi ditambahkan ke database Developmental Disorder Gene to Phenotype (DDG2P).
Selain itu, ketika diterapkan pada mutasi missense (DNM) de novo, model ini menandai 7% varian dalam kasus sebagai kasus yang parah, dibandingkan dengan hanya 0,5% pada kontrol yang sehat, yang menunjukkan tingkat pemisahan yang tinggi antara yang patogen dan yang jinak. variasi.
Debora Marks, Profesor Biologi Sistem di Harvard Medical School, menekankan bahwa alat ini dirancang untuk menerjemahkan perolehan statistik ini menjadi hasil klinis yang nyata. “Tujuan kami adalah mengembangkan model yang mengurutkan varian berdasarkan tingkat keparahan penyakit – memberikan pandangan genom seseorang yang diprioritaskan dan bermakna secara klinis.”
Mengkalibrasi Proteome
Model canggih sebelumnya, termasuk EVE dan AlphaMissense, unggul dalam memberi peringkat varian dalam satu gen tetapi kesulitan membandingkan tingkat keparahan di berbagai gen. Akibatnya, skor tinggi sering kali muncul untuk varian yang mengganggu fungsi protein namun belum tentu menyebabkan penyakit parah dalam konteks manusia.
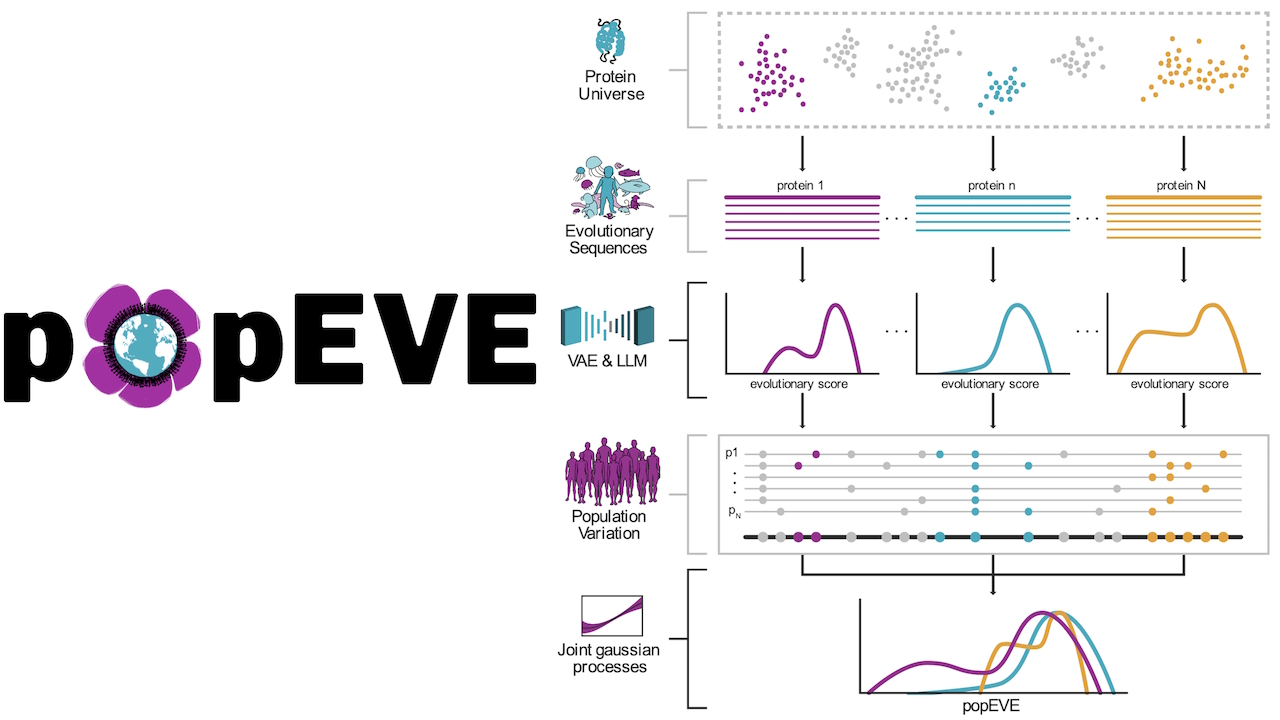
popEVE memecahkan masalah ini dengan menggabungkan data evolusi mendalam (menggunakan model bahasa EVE dan ESM-1v) dengan batasan populasi manusia. Untuk menentukan varian yang dapat ditoleransi secara alami, tim menggunakan data dari UK Biobank (UKBB) dan gnomAD v2.
Proses Gaussian laten digunakan untuk mengkalibrasi skor evolusi terhadap variasi manusia yang diamati, sehingga menciptakan skor “kerusakan” terpadu. Melalui penyesuaian ini, terobosan klinis besar menjadi mungkin dilakukan: analisis “tunggal”, di mana varian penyebab dapat diprioritaskan hanya dengan menggunakan eksome anak.
Metode tradisional biasanya memerlukan pengurutan “trio”(orang tua + anak) untuk mengidentifikasi mutasi de novo, sebuah proses yang sering kali sangat mahal atau tidak mungkin dilakukan secara logistik.
Mafalda Dias, peneliti di Center for Genomic Regulation, menyoroti implikasi praktis dari kemampuan ini. “Klinik tidak selalu memiliki akses ke DNA orang tua dan banyak pasien datang sendiri. popEVE dapat membantu para dokter ini mengidentifikasi mutasi penyebab penyakit.”
Challenging AlphaMissense
AlphaMissense Google DeepMind, yang dirilis pada September 2023, sebelumnya menetapkan standar baru dengan mengkategorikan 89% dari semua kemungkinan varian missense. Namun, tim Harvard berpendapat bahwa meskipun AlphaMissense akurat untuk stabilitas protein, ia tidak memiliki kalibrasi klinis yang diperlukan untuk diagnosis.
Analisis statistik menunjukkan AlphaMissense memperkirakan rata-rata lima varian “patogen” per rata-rata orang, sedangkan popEVE memperkirakan kurang dari satu. Perbedaan tersebut sangat penting dalam situasi klinis, karena prediksi yang berlebihan dapat menyebabkan kesalahan diagnosis dan kecemasan yang tidak perlu.
Makalah PrpopEVE lebih lanjut mencatat:
“popEVE mengidentifikasi 442 gen dalam kohort gangguan perkembangan, termasuk bukti dari 123 kandidat baru, banyak di antaranya yang tidak memerlukan pengayaan kohort secara luas.”
“Akhirnya, kami menunjukkan bahwa temuan ini dapat direproduksi dari analisis pasien melakukan eksom sendiri, menunjukkan bahwa popEVE memberikan jalan baru untuk analisis genetik dalam situasi di mana metode tradisional gagal.”
Meskipun ada peningkatan kinerja, popEVE tetap menjadi alat penelitian dan belum menerima izin FDA untuk digunakan sebagai perangkat diagnostik mandiri. Marks Lab menyediakan model ini melalui portal popEVE dan repositori popEVE, yang kontras dengan sifat alat kesehatan AI komersial yang sering kali merupakan hak milik.
Aplikasi di masa depan tidak hanya sekedar diagnosis hingga penemuan obat, karena model ini dapat menentukan patogen tertentu mekanisme dalam struktur protein.
Rose Orenbuch, peneliti di Marks Lab, menyatakan optimisme tentang integrasi alat ini ke dalam alur kerja klinis. “Saya merasa kita selangkah lebih dekat dengan popEVE yang berguna dalam upaya sehari-hari untuk mendiagnosis penyakit genetik dengan lebih cepat.”